成都生物所在基于对比学习的MS/MS谱图-结构预训练模型鉴定代谢物研究中获新进展
来源:生物资源利用中心
作者:夏兵
时间:2024-10-16
代谢物是在代谢过程中化学转化的小分子,提供了细胞状态的直接读数。阐明代谢物的结构是代谢组学的首要研究任务,在药物研发、生物标志物发现、天然产物研究等方面具有重要意义。液相色谱-串联质谱(LC-MS/MS)是该领域广泛使用的分析手段。在代谢物研究中,对代谢物串联质谱(MS/MS)数据的结构注释是一项关键挑战。人工智能(AI)技术已彻底改变了质谱数据的解析方式,促进了代谢组学领域中“暗物质”的识别。现有的创新方法主要侧重于将MS/MS谱图或分子结构转化为统一模式,以实现基于相似性的比较和解析。对比学习(Contrastive learning)是一种用于学习表征的无监督学习方法,可以将不同的模态嵌入到一个共同的潜在空间中,以便直接进行比较。
中国科学院成都生物研究所生物资源利用中心周燕研究团队开发了一种新颖的MS/MS谱图-分子结构预训练对比学习模型(Contrastive Mass Spectra-Structure Pretraining Model,CMSSP),该模型的主要目标是建立一个表示空间,以实现MS/MS谱图与分子结构间的直接比较,从而超越不同模态的限制。该模型在两个基准测试集上的评估结果显著超越了之前的先进方法,在CASMI 2017数据集上的top-1命中率提高30%。此外,该模型在七个化学类别中都表现出的优越的鉴定准确性,证明了其稳健性和通用性。实际应用表明,对Glycyrrhiza glabra中30个代谢物的MS/MS数据进行注释,其top-1和top-3的鉴定准确率分别达到86.7%和100%。CMSSP模型是解析和解释复杂MS/MS数据的强大工具,不仅增强了代谢组学的分析能力,也为理解复杂生物系统提供有力支持。
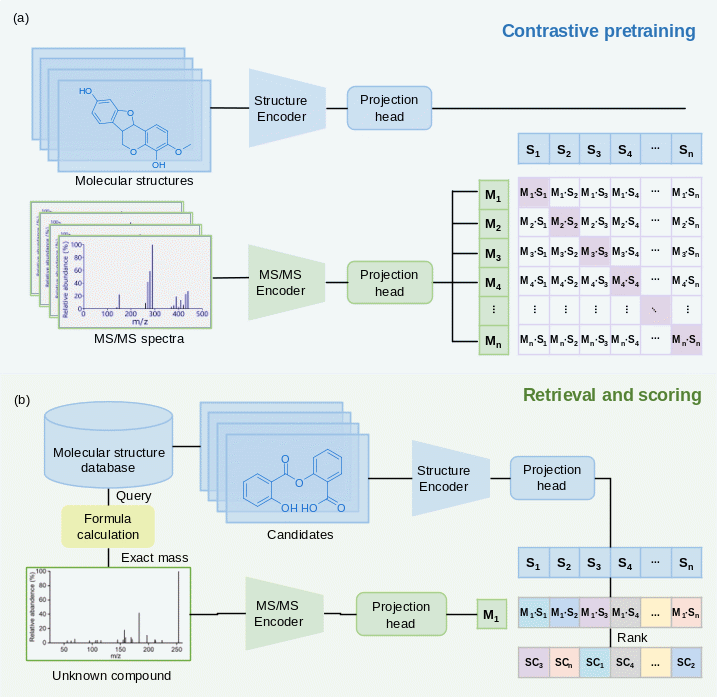
图1.CMSSP的工作流程示意图
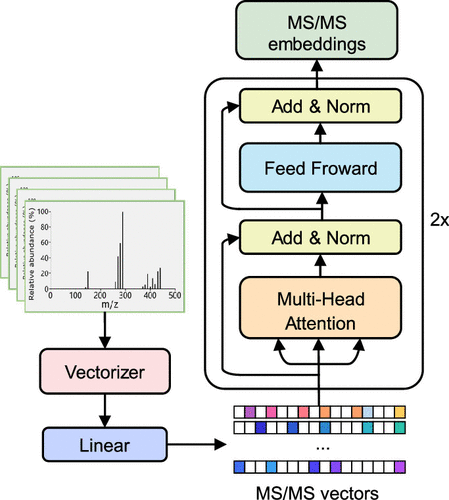
图2.MS/MS编码器结构示意图
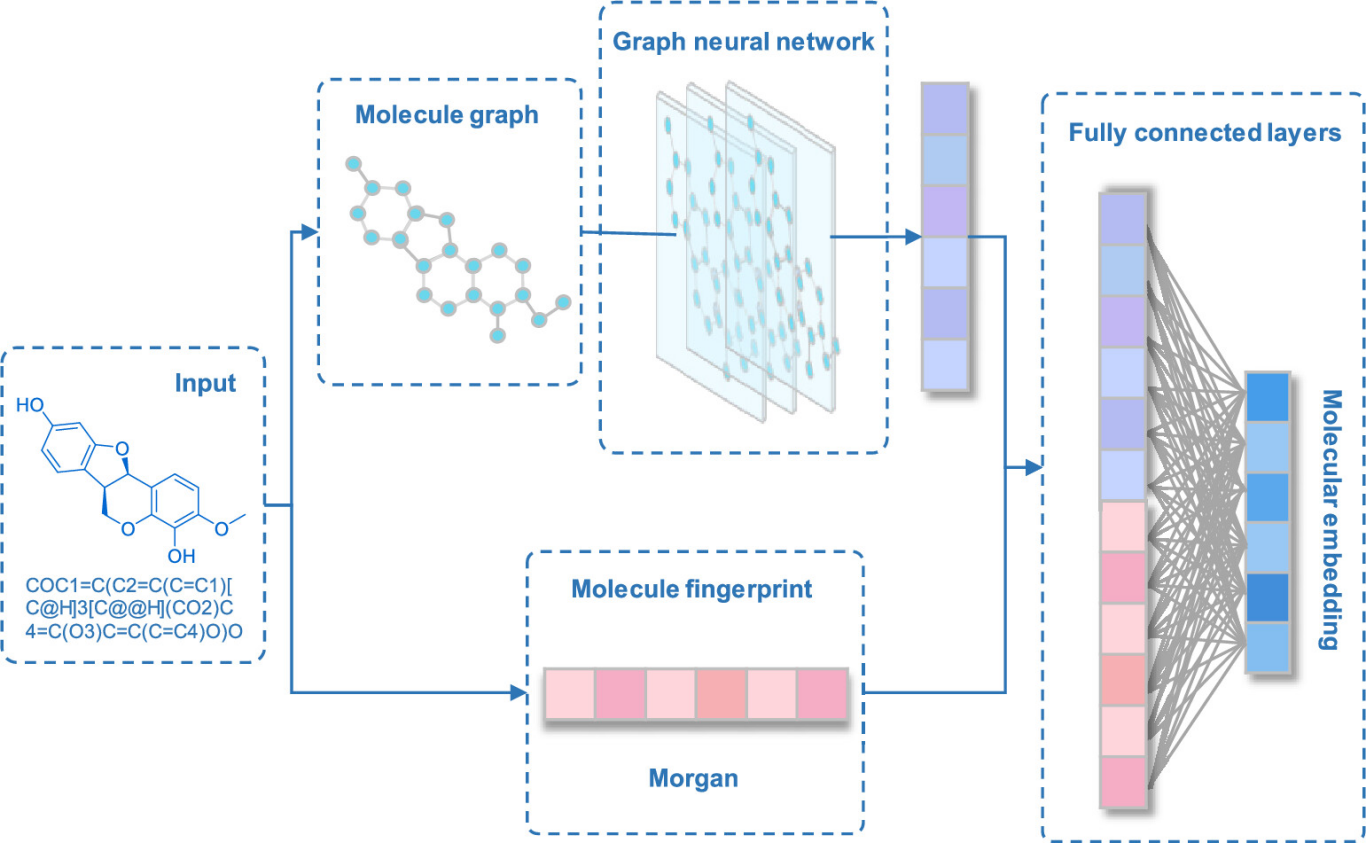
图3.分子结构编码器结构示意图
上述研究结果于2024年10月14日以“CMSSP: A Contrastive Mass Spectra-Structure Pretraining Model for Metabolite Identification”为题发表在分析化学国际顶级期刊Analytical Chemistry上。成都生物研究所博士研究生陈璐和夏兵青年研究员为本文共同第一作者,通讯作者为成都生物研究所周燕研究员和夏兵青年研究员。本研究得到了四川省重点研发项目(No. 2022YFS0511)、先正达(Syngenta)博生生项目(SPF146)和成渝区域中心交叉融合项目(No. 90E3C305)的资助。




