成都生物所在超大基因组转座子动态研究中取得进展
作者:王杰
时间:2021-08-18
真核生物“流行”小基因组,绝大多数种类的基因组小于 4 Gb,然而它们基因组大小的差异竟达到惊人的 64,000 倍,八倍体百合日本重楼Paris japonica的基因组最大(150 Gb),二倍体石花肺鱼Protopterus aethiopicus的基因组次之(130 Gb),原生动物微孢子虫Encephalitozoon intestinalis的基因组最小(2.3 Mb)。真核生物单倍体基因组中DNA的含量与其进化上的地位或者说复杂性无关甚至矛盾的现象被称谓“C 值悖论”(C-value paradox or enigma)。植物基因组大小的快速变化可以通过多倍化实现,然后经过大规模的染色体重排和删除事件将基因组的扩张稳定下来。然而,对动物而言,基因组的多倍化现象比较罕见,基因组大小的进化之谜令人费解。“为什么一些基因组很大,另一些又非常小”也因此被 Science杂志列为当今世界最前沿的 125 个科学问题之一。
转座子(transposable elements, TE)可在染色体DNA上自主复制和“跳跃”,曾长期被认为是 “垃圾信息”, 近年来被证实是驱动基因组扩张的主要组分。超大基因组(一般指基因组大于10 Gb)在动物中非常罕见,我们以两栖动物原始类群版纳鱼螈(Ichthyophis bannanicus)为研究对象(基因组大小约12.2G),采用低覆盖度(0.1×)测序研究其转座子的景观和动态。这是两栖纲蚓螈目第一个基因组层面的解析。
该研究发现,重复序列占鱼螈基因组的78%,而转座子约占68%,包含至少25个超家族,以DIRS和Jockey转座子最为丰富,分别占基因组的30.2%和20.6%,这样的组成与两栖类蝾螈目和无尾目甚为不同,而与“四足动物最亲近的鱼类活化石”—— 肺鱼更为接近,提示转座子在四足动物祖先由水登陆过程中起重要作用。DIRS和Gypsy转座子被易位重组介导的删除速率与其它脊椎动物相似,都比较低,并且转座子的大多数超家族依然活跃,表达量占体细胞总表达量的15%,表明超大基因组在历史扩张中保持了比较高的转座子多样性。
此外,我们归纳总结了文献中有关转座子数量与多样性的动态模型,并首次引入生态学中的Shannon与Gini-Simpson指数对鱼螈及其它10种模式动物转座子的多样性进行同样的计算分析,以验证这些模型是否符合实证。据此提出今后应将此类一致性分析引入类似的研究中,以便反映转座子与宿主博弈的规律。
研究结果以“Gigantic Genomes Provide Empirical Tests of Transposable Element Dynamics Models” 为题,发表于遗传学领域top期刊Genomics Proteomics Bioinformatics (IF = 7.691,https://doi.org/10.1016/j.gpb.2020.11.005 )。中科院成都生物所江建平研究组王杰副研究员为论文第一作者和共同通讯作者,江建平研究员、李家堂研究员、龚宇舟博士,中南财经政法大学信息与安全工程学院王会举教授、中国农业科学院蜜蜂研究所孙成研究员为合作作者,科罗拉多州立大学Rachel Lockridge Mueller教授为论文共同通讯作者。该研究得到国家自然科学基金面上项目(31570391)、国家重点研发计划“珍稀动物濒危机制及保护技术研究”(2016YFC0503200)等项目资助。

图1四肢退化消失、营穴居生活的国家二级重点保护动物—版纳鱼螈(任金龙拍摄)
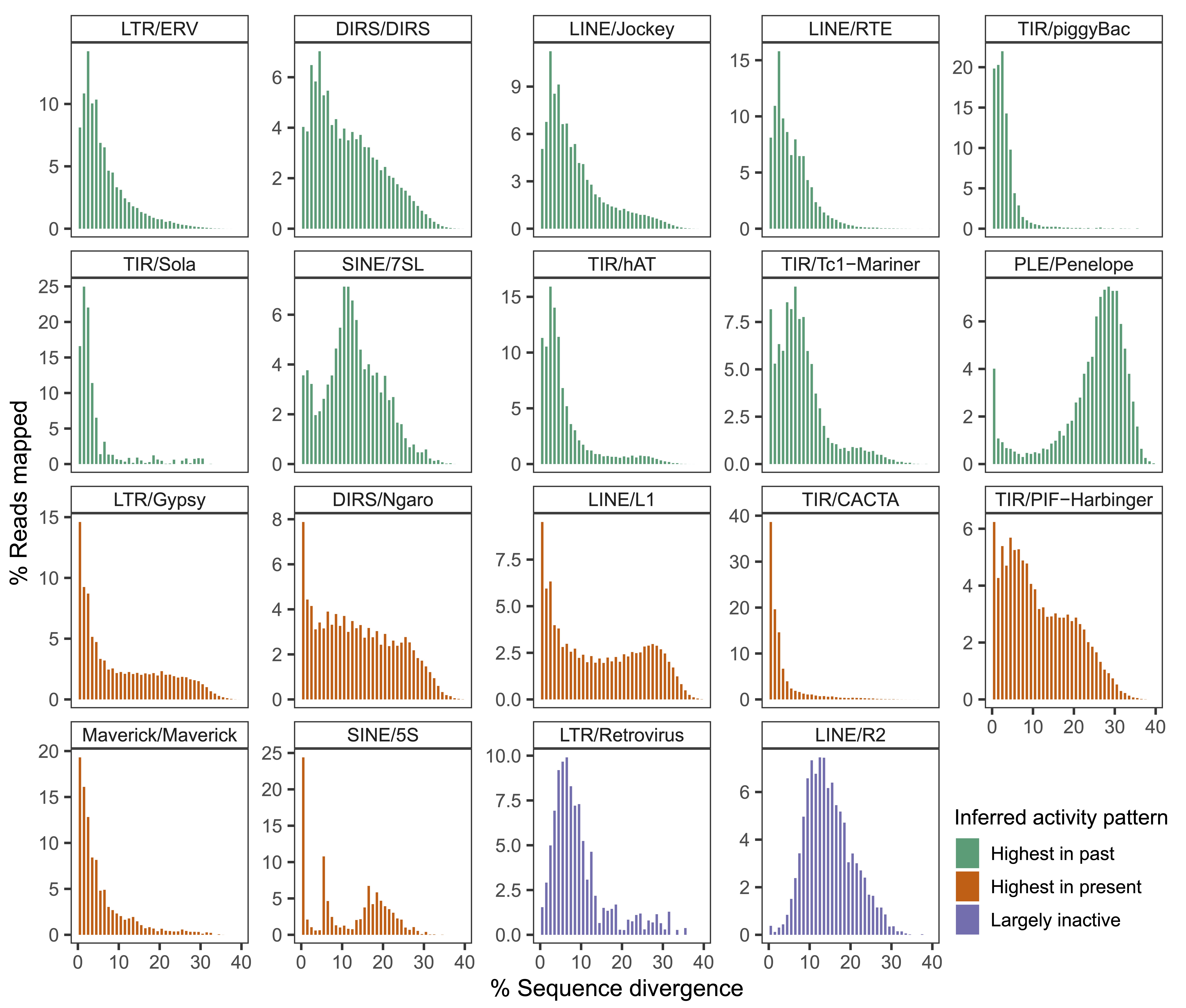
图2 版纳鱼螈基因组中19个超家族转座子的历史动态
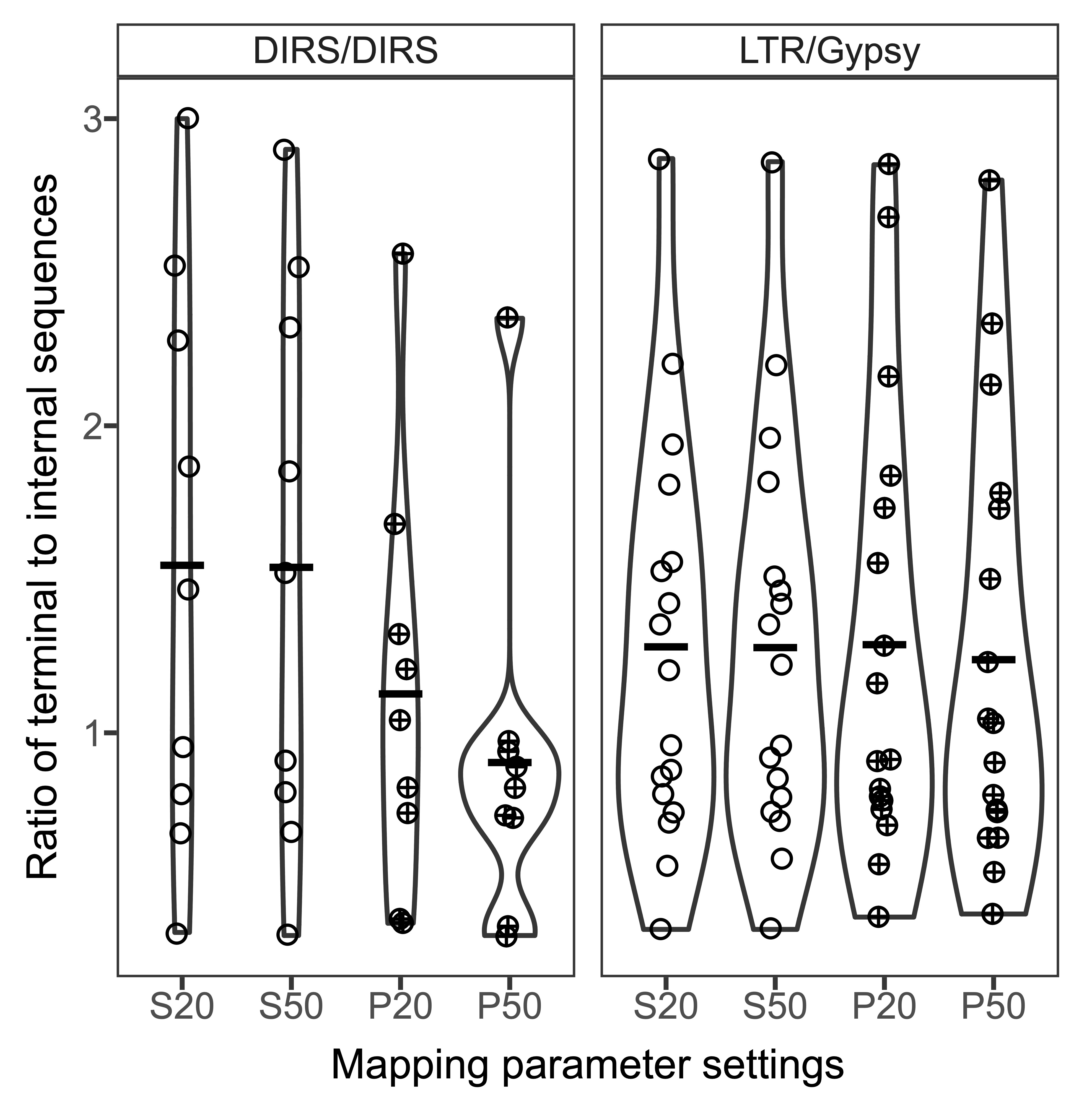
图3 DIRS与Gypsy超家族转座子由移位重组引起的删除率比较
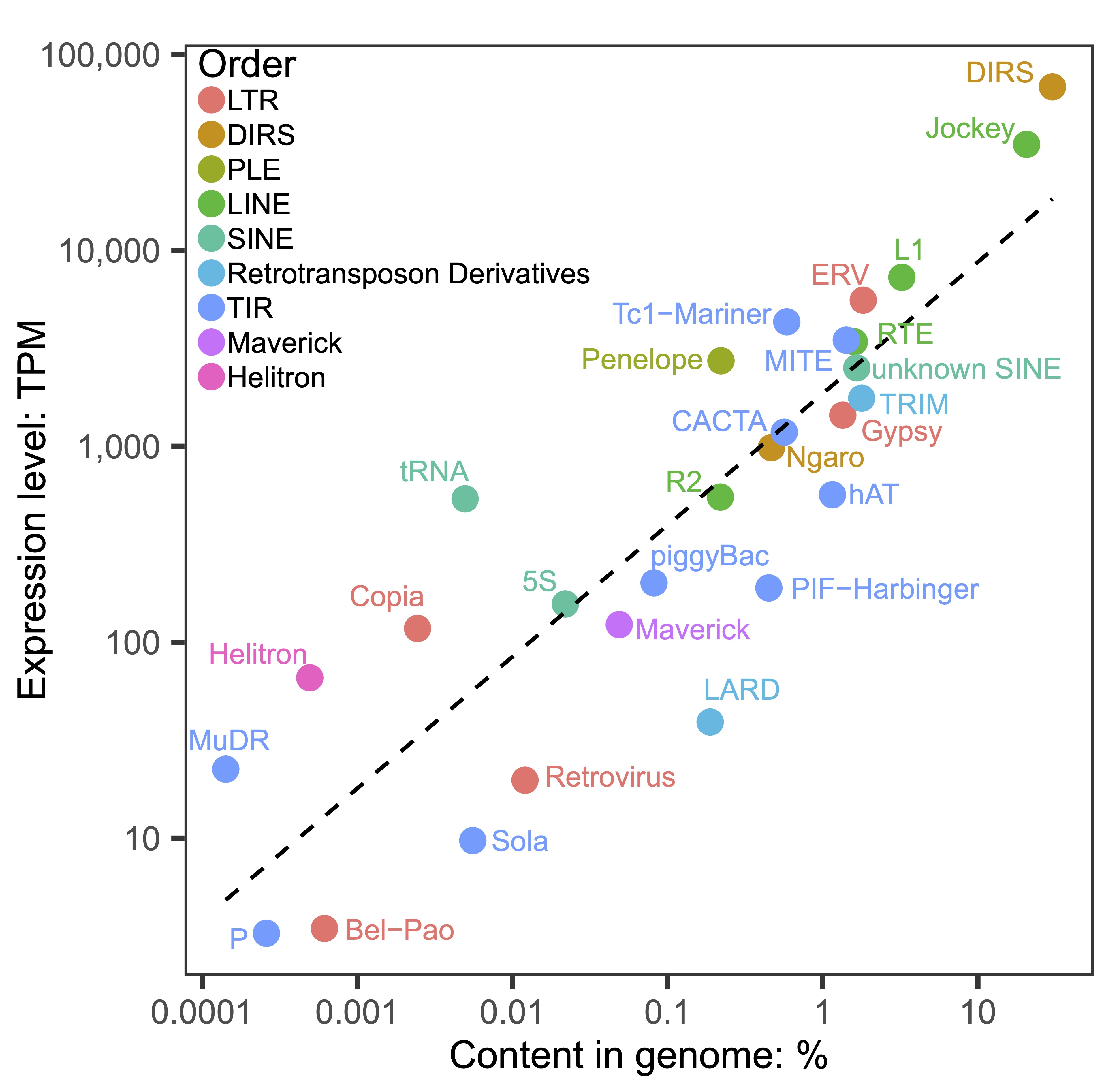
图4 不同超家族转座子的表达与其在基因组中的含量呈线性相关(R = 0.879, P < 0.001)
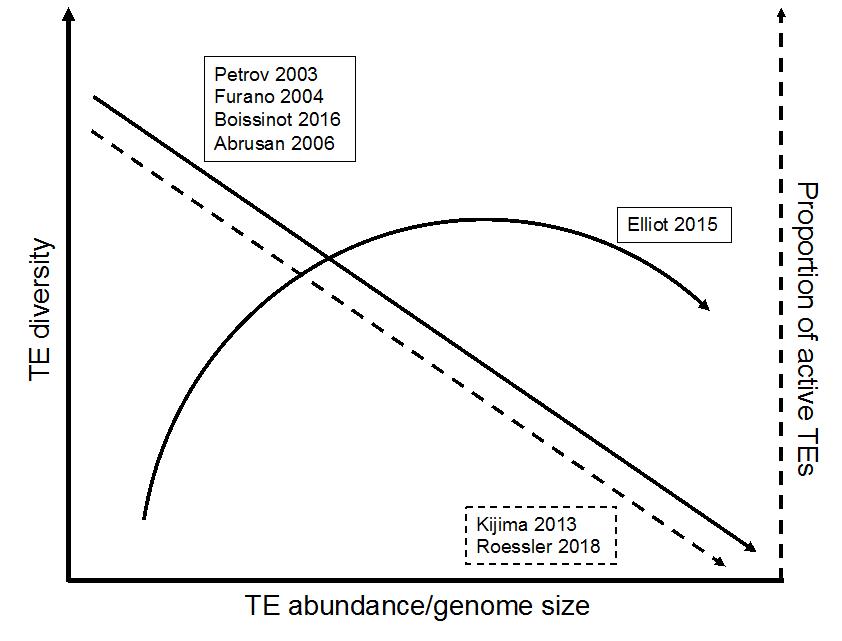
图5 转座子随基因组大小变化的动态模型




