成都生物所在麻蜥属胎生类群DNA条形码研究取得新进展,确认准噶尔麻蜥在中国西北有分布
作者:刘金龙,郭宪光
时间:2021-04-23
随着分析技术的不断发展,DNA条形码已被广泛应用于物种界定和发现、甚至全球生物多样性的评估[1]。DNA条形码对于具有较大的形态保守性或模糊性的类群,进行分子系统发育推论和分类学研究特别有益。例如,最近利用DNA条形码,在茧蜂科(Braconidae)的11个亚科中界定了超过400个新物种[2]。尽管DNA条形码在进化和分类学研究中有其固有的缺陷,而从中推断出的证据可以指导后续有针对性地开展形态学研究,并利用来自多基因位点、地理分布和生态学等多重证据,对物种边界做出更可靠的推断[3]。
麻蜥属(Eremias)中的胎生类群,即密点-荒漠麻蜥复合体,是分类复杂的著名代表。该复合体存在复杂的种内形态变异和种间相似性,物种界定比较困难;历经了漫长的分类学修订,至今有多达18个种/亚种被提出,其中最近被描记的一个新物种就是准噶尔麻蜥(Eremias dzungarica Orlova et al., 2017)[4]。该种与同属近缘物种相比,具有明显的遗传和形态分化,被报道仅分布于蒙古西部阿勒泰山周边较高海拔区域(2400?2600 m)的岩石丘陵和砾石峡谷生境、以及哈萨克斯坦东部的低海拔(400-1000 m)沙漠地带。因此,处在蒙古西部和哈萨克斯坦东部之间的中国新疆西北部是否有该新种的分布,就不得而知。此外,Orlova等(2017)首次利用DNA条形码数据来解析密点-荒漠麻蜥复合体种群遗传分化和系统发育关系,并基于线粒体基因谱系、形态以及地理分布数据进行初步的物种推测和界定。然而,他们尚未利用更严格的条形码技术来深入探究此复合体中的谱系遗传距离的分布模式,进而有效地验证物种边界。
为了厘清这些问题,成都生物所两栖爬行动物多样性与保护项目组刘金龙博士后在郭宪光副研究员的指导下,与哈萨克斯坦教科部动物研究所合作,获取和整合DNA条形码数据,解析了密点-荒漠麻蜥复合体内种间和种内的遗传距离分布式样,并结合物种界定分析,对此复合体内的物种多样性及其成因进行了探讨;利用整合分类学的方法(即利用多学科之间的互补性,将来自不同学科领域的新理论和方法整合为一体的一种分类方法[5]),明确了准噶尔麻蜥在中国新疆阿勒泰地区有分布;通过形态学研究也揭示了较多与该物种的原始描述不一致之处。首先,通过多年的野外科考和样本采集,在哈萨克斯坦东部和毗邻的中国阿勒泰地区采集了30号先前被归为密点麻蜥的标本,对DNA条形码COI基因进行测序,与Orlova等人的条形码数据进行整合分析。我们检测到此复合体不存在条形码间隙,也就是说,种内和种间遗传距离分布有相互重叠;结合物种界定分析的结果,暗示出在有较高种内多样性的Eremias sp. 3中有潜在的隐存种,并且在支系A中有多个新近演化的物种。其次,通过分子系统发育和形态学检查,该研究采集的30只个体均被可靠地鉴定为准噶尔麻蜥,证实了该物种在中国新疆阿勒泰地区有分布。由于该研究的样本量较大,形态学分析与准噶尔麻蜥原始描述存在许多不一致之处,主要表现为性二态性以及不同性状的量值范围更宽。
准噶尔麻蜥在蒙古与哈萨克斯坦的分布原以为是断开的,该研究确认其在中国新疆阿勒泰地区有分布,这一发现使其分布区相连。准噶尔麻蜥是新疆爬行动物新纪录种,也是中国爬行动物新纪录种,为干旱荒漠区生物多样性保护提供了重要的基础信息。该研究也暗示麻蜥属胎生类群多样性被低估,有潜在的隐存种,为进一步探讨麻蜥属多样性与进化提供了线索与思路。研究成果以Does the Dzungarian racerunner (Eremias dzungarica Orlova, Poyarkov, Chirikova, Nazarov, Munkhbaatar, Munkhbayar & Terbish, 2017) occur in China? Species delimitation and identification with DNA barcoding and morphometric analyses为题发表在动物学Top杂志Zoological Research [2021, 42(3): 287-293]。该研究得到了中科院A类战略性先导科技专项、国家自然科学基金的共同资助。
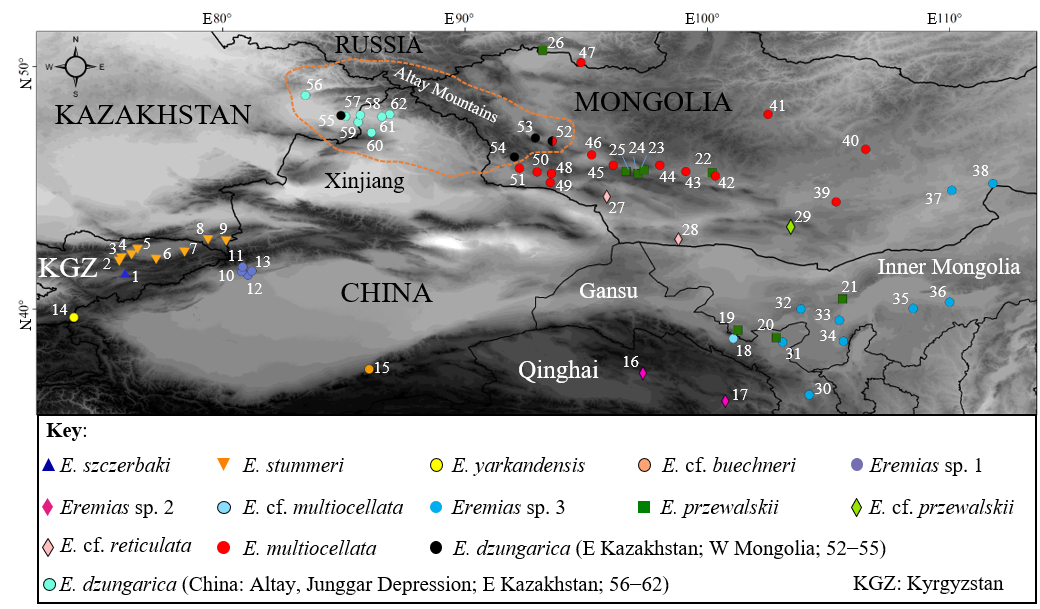
图1 密点-荒漠麻蜥复合体样点,橙色虚线显示准噶尔麻蜥的分布范围
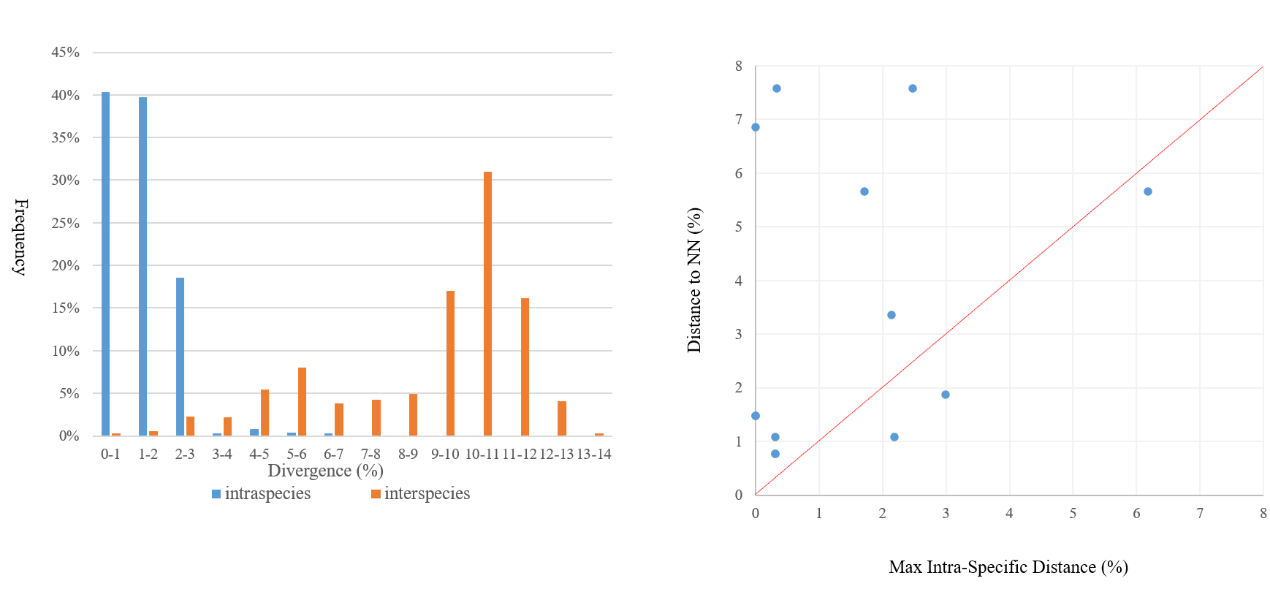
图2 密点-荒漠麻蜥复合体在种内和种间分类水平上遗传距离的频率分布(左)以及各个物种最大种内变异和最小遗传距离的散点图(右)
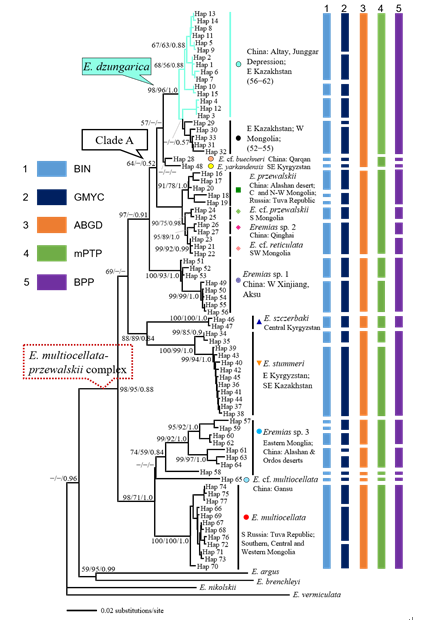
图3 基于DNA条形码COI基因序列构建的系统发育树及物种界定分析

图4哈萨克斯坦东部和中国阿勒泰西部地区的准噶尔麻蜥生态照片(A1-A5:雌;B1-B3: 雄)
[1] DeSalle R, Goldstein P. 2019. Review and interpretation of trends in DNA barcoding. Frontiers in Ecology and Evolution, 7: 302.
[2] Sharkey MJ, Janzen DH, Hallwachs W, Chapman EG, Smith MA, Dapkey T, et al. 2021. Minimalist revision and description of 403 new species in 11 subfamilies of Costa Rican braconid parasitoid wasps, including host records for 219 species. ZooKeys, 1013: 1-665.
[3] Damm S, Schierwater B, Hadrys H. 2010. An integrative approach to species discovery in odonates: from character-based DNA barcoding to ecology. Molecular Ecology, 19(18): 3881-3893.
[4] Orlova VF, Poyarkov Jr NA, Chirikova MA, Nazarov RA, Munkhbaatar M, Munkhbayar K, et al. 2017. MtDNA differentiation and taxonomy of Central Asian racerunners of Eremias multiocellata-E. przewalskii species complex (Squamata, Lacertidae). Zootaxa, 4282(1): 1-42.
[5] Will KW, Mishler BD, Wheeler QD. 2005. The perils of DNA barcoding and the need for integrative taxonomy. Systematic Biology, 54(5): 844-851.




