
最近,瑞士日内瓦大学(UNIGE)的一个研究小组,构建了一个大的数据库,其中就包括一种爬行动物——玉米蛇的新基因组测序,这种蛇越来越多地被用来了解爬行动物的进化。在同一个实验室,研究人员发现了导致这一物种白化现象(albinism)的确切变异,相关研究结果发表在十一月二十四日的《Scientific Reports》。
基因组学可以让我们更好地探讨生命世界的演化。事实上,描述每一个基因的功能,可让我们能够了解蛇如何失去肢体,或者各种皮肤色彩如何演变。不幸的是,爬行动物很少出现在基因组大数据库中。鉴于此,UNIGE科学学院遗传和进化系的Athanasia Tzika博士,构建了一个数据库,包括来自主要爬行动物演化谱系的基因组序列,该数据库称为Reptilian Transcriptomes Database 2.0。Athanasia Tzika说:“我们的目标是,通过对来自几个爬行动物物种的所有基因进行测序,产生很大一部分的缺失数据。为了达到这个目标,我们使用表达最大数目基因的组织,如大脑和肾脏。”其他多个研究团队也产生了测序数据,但每一个研究小都用不同的方法进行数据分析,从而使爬行动物基因组进化的研究变得非常困难。因此,Athanasia Tzika工作的另一部分,是收集这些数据并开发一种生物信息学方法,来产生一个统一的数据库,可让研究人员自由访问和定期更新。这一工具对于世界各地致力于脊椎动物(特别是爬行动物)发育和进化的研究人员,将是非常有用的。
玉米蛇,一种新的模式生物
基因组测序是一项巨大的任务。它需要确定被研究动物的全基因组序列,对应于玉米蛇的20亿个核苷酸。DNA包含四个基本的分子(核苷酸)——ACGT,通过配对组装(A与T,C与G),并构成了每个基因的序列。要分析基因组,研究人员必须读取100到250个核苷酸的DNA短片段,然后再组装以重建形成每个染色体的巨大链。
如今,科学家们可以进行这样的组装,建立约一百万个核苷酸的片段。这项工作已经由UNIGE遗传和进化系的Michel Milinkovitch教授带领的团队完成。Athanasia Tzika解释说:“我们的目的是要获得一个人们可以依赖的真正的爬行动物基因组模型。在这里,我们覆盖了蛇全基因组大小的大约85%。”这项工作的共同作者Asier Ullate解释说:“还有很多额外的工作要做,因为,为了获得与人类基因组质量相似的测序,我们需要组装的染色体,可能形成2亿多个核苷酸。对于研究爬行动物的发育和进化来说,玉米蛇是非常完美的,因为它很容易繁育,它是卵生的,并且是无毒的。本物种也可用于研究遗传决定论和皮肤颜色的演化,对于那些以自然栖息地作为伪装的动物,这尤为重要。我们很高兴地宣布,玉米蛇也是有限的模型物种组中的一个成员,可允许研究人员更好地了解造成物种进化多样性和复杂性的生物和物理机制。”
寻找引起白化现象的突变
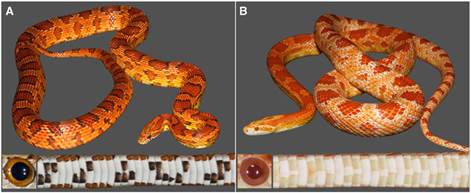
在同一UNIGE实验室中,Suzanne Saenko与瑞典一个研究小组合作,在玉米蛇中确定负责白化现象(由于黑色素产生缺陷而引起的一种白化病)的突变。野生型玉米蛇的皮肤呈浅橙色背景颜色,上面覆盖着暗橙色鞍纹,鞍纹的周围是宽大厚实的黑纹。然而,有些玉米蛇并不符合这种标准的形态:它们在皮肤和眼睛中缺乏黑色素的所有迹象。瑞士的研究团队决定搜索决定特定颜色的基因突变。为此,他们将野生型玉米蛇与白化个体进行杂交,并对繁育的杂交后代进行测序。Milinkovitch解释说:“由于大量的测序数据,我们确定了出故障的基因。”这个基因被称为OCA2,编码位于细胞内隔室(称为黑素体,含有黑色素)膜中的受体。该受体控制使黑色素合成的酸度适当水平。
新测序的玉米蛇基因组,将大大协助研究人员准确识别引起多个蛇皮肤着色差异的其他突变。这将使我们在未来研究中能够节省大量的时间。因为在白化现象中,这些自发突变——可能随机出现在任何个体中,是自然选择作用于进化的基本刺激因素。没有突变,遗传变异和新奇产物——解释新物种的进化,是不可能存在的。UNIGE的研究人员将尝试了解玉米蛇表现出色彩图案很大改变(如纵向线而不是横向鞍纹)的一些突变,从而为在具有不同肤色图案的物种(如老虎和豹)之间进行比较,开辟了新的途径。(来源:生物通 王英)
Amelanism in the corn snake is associated with the insertion of an LTR-retrotransposon in the OCA2 gene
Abstract The corn snake (Pantherophis guttatus) is a new model species particularly appropriate for investigating the processes generating colours in reptiles because numerous colour and pattern mutants have been isolated in the last five decades. Using our captive-bred colony of corn snakes, transcriptomic and genomic next-generation sequencing, exome assembly, and genotyping of SNPs in multiple families, we delimit the genomic interval bearing the causal mutation of amelanism, the oldest colour variant observed in that species. Proceeding with sequencing the candidate gene OCA2 in the uncovered genomic interval, we identify that the insertion of an LTR-retrotransposon in its 11th intron results in a considerable truncation of the p protein and likely constitutes the causal mutation of amelanism in corn snakes. As amelanistic snakes exhibit white, instead of black, borders around an otherwise normal pattern of dorsal orange saddles and lateral blotches, our results indicate that melanocytes lacking melanin are able to participate to the normal patterning of other colours in the skin. In combination with research in the zebrafish, this work opens the perspective of using corn snake colour and pattern variants to investigate the generative processes of skin colour patterning shared among major vertebrate lineages.
原文链接:http://www.nature.com/articles/srep17118



